Quiero hacer un mapa de clúster / mapa de calor de datos de presencia y ausencia de genes de pacientes donde los genes se agruparán en categorías (por ejemplo, quimiotaxis, endotoxina, etc.) y se etiquetarán adecuadamente. No he encontrado ninguna opción de este tipo en la documentación marina. Sé cómo generar el mapa de calor, pero no sé cómo etiquetar yticks como categorías. Aquí hay una muestra (no relacionada con mi trabajo) de lo que quiero lograr:
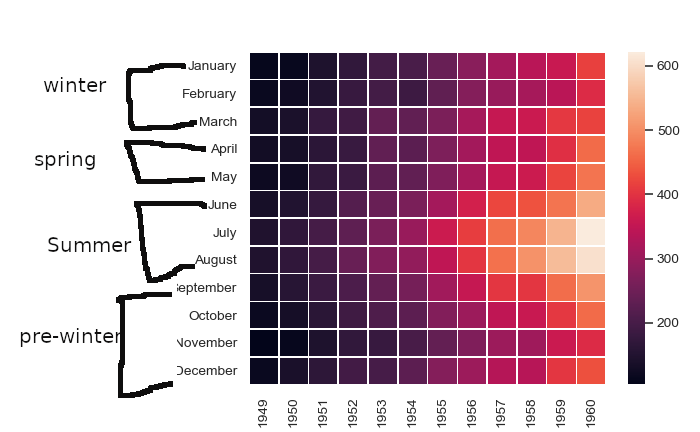
Aquí, las etiquetas de enero, febrero y marzo reciben etiquetas de grupo de invierno y otras etiquetas de etiqueta también tienen etiquetas similares.

Respuestas:
He reproducido el ejemplo que diste en seaborn, adaptando la respuesta de @ Stein desde aquí .
Da:
Espero que ayude.
fuente
Todavía no he probado esto con seaborn, pero lo siguiente funciona con matplotlib de vainilla.
fuente